Rによる機械学習:caretパッケージの使い方
この記事では、R言語を使って機械学習法を効率的に実装することのできるパッケージ「caret」について説明します。
ニューラルネットワークにSVM、ランダムフォレストに勾配ブースティング……。機械学習には実に様々な手法があります。
これらの手法を実装するにあたって、R言語には様々なパッケージが用意されています。自分で一から計算するのと比べると大変に簡単です。
しかし、たくさんのパッケージを管理し、それらの使い方を逐一覚えるというのもなかなか大変ですね。
そこで登場するのがパッケージ「caret」。
caretとは、数々の機械学習関連のパッケージたちを統一的に取り扱うためのパッケージです。
この記事では、caretパッケージの使い方を説明したうえで、ニューラルネットワーク、ランダムフォレスト、そしてXgboostを使った勾配ブースティングを実装してみます。
ソースコードはすべてこちらに置いてあります。
新規作成:2016年11月05日
最終更新:2016年04月24日
スポンサードリンク
目次
- caretパッケージとは
- caretパッケージでよく使う関数
- 機械学習法を使って予測を行う手順
- train関数の使い方
- 予測の方法
- caretを使った回帰の例
- caretを使った分類の例
- caretを使って勾配ブースティング(Xgboost)
1.caretパッケージとは
caretは、数々の機械学習関連のパッケージたちを統一的に取り扱うためのパッケージです。
なお、機械学習のことをまったく知らないという方は、先にこちらの入門記事を参照してください。
caretパッケージを一つだけインストールすれば、ほかのパッケージは何もいりません。
ニューラルネットワークを計算するパッケージも、SVMを作成するパッケージも、みんなみんなcaretパッケージが管理してくれます。私たちはcaretパッケージの操作を覚えるだけで十分です。
便利ですね。
caretパッケージを使うと、以下のことができます。
- 機械学習法を用いた予測モデルの作成
- ハイパーパラメタのチューニング
- 作成されたモデルを使った予測
少し見慣れぬ用語が出てきたかもしれません。
caretパッケージの使い方を学ぶ前に、機械学習法による予測の手順を先に説明します。
2.機械学習法を使って予測を行う手順
機械学習法を使って予測するには、以下の手順を踏みます。
- 使用する手法を決める
- その手法を実装する方法を決める
- パラメタのチューニングをする
- モデルを使って予測する
- 予測の評価をする
1と4に関しては、忘れる人はあまりいないのですが、2と3、5はスルーしやすいので注意が必要です。
順番に説明します。
1.使用する手法を決める
一口に機械学習法といっても様々な手法があります。
手法とは、例えばニューラルネットワークであったりランダムフォレストであったりします。
まずは、どの手法を使うかを決めます。
手法を決める明確な基準は実はなくって、いくつか候補を選んで、それを全部試すということもしばしばです。
2.その手法を実装する方法を決める
例えばランダムフォレストを使うと決めたとしましょう。
ランダムフォレストをRで実装する方法は、実はいくつかあります。
自分でイチから作るというのも一つでしょうが、多くの場合はRのパッケージを使うと思います。
で、実は、同じランダムフォレストを計算する機能を持ったパッケージが複数あるんですね。
ランダムフォレストを計算するパッケージは例えば「randomForest」パッケージであったり「ranger」パッケージであったり「Rborist」なんてものあります。
どれも一長一短で、何を使うのが良いか、悩ましいですね。
で、結局いろんなパッケージで試してみて、一番いいのを選ぶことになります。
3.パラメタのチューニング
パラメタのチューニングが、機械学習における一つの鬼門です。
機械学習は名前の通り「機械が勝手に学習してくれる手法」なのですが、全自動というわけにはいきません。
ある程度は人間が決めてやらなくてはならない部分があるのです。
人間があらかじめ与えておく必要のあるパラメタのことを「ハイパーパラメタ」と呼びます。
例えばニューラルネットワークだと、中間層にあるニューロンの個数はあらかじめ与えておく必要があります。
ニューロンの個数はいくつが良いでしょうか。2個? 3個?
そんなものはわからないので、いくつかの候補を試してみて、最も予測精度が良いものを選びます。
なお、チューニングすべきパラメタが1つとは限りません。
ニューラルネットワークの場合は、「中間層のニューロンの個数」と「手持ちのデータにどこまで合わせるか」という、手持ちのデータへの依存度のようなパラメタもあって、そいつもチューニングしなければなりません。
2つ以上のパラメタをチューニングする場合は、グリッドサーチと呼ばれる手法を使うのが普通です。
具体的には、以下のように、対にしてパラメタを変えていきます。
| ニューロンの個数 | データへの依存度 |
|---|---|
| 1 | 1 |
| 1 | 0.5 |
| 1 | 0.01 |
| 2 | 1 |
| 2 | 0.5 |
| 2 | 0.01 |
ニューロンの個数を2パタン。
データへの依存度を3パタン用意すると、2×3=6パタンも試さなければなりません。
大変に面倒ですが、このハイパーパラメタを雑に設定すると予測精度が落ちてしまうこともよくあります。
めんどくさいですが、なるべくやったほうがいいです。
4.モデルを使って予測する
これは簡単。単に予測をするだけです。
パッケージを使っていれば、たいていは予測用の関数が用意されています。
5.予測の評価をする
最後は評価です。
評価をすることによって、
・どの手法の
・どのパッケージの
・どのパラメタを
採用すべきかを判断します。
で、一番予測精度がいいやつを使って予測することになります。
たくさんの手法、たくさんのパッケージ、たくさんのパラメタの組み合わせをしらみつぶしに評価する。
普通にこれをやろうと思うと、気が遠くなってしまいますね。
そこで登場するのがパッケージ「caret」。
caretパッケージは、様々な手法・パッケージを統一的に取り扱うことができ、パラメタのチューニングもほとんど自動でやってのけます。
次からは、caretパッケージの使い方を学びます。
3.caretパッケージでよく使う関数
たくさんの関数があるのですが、以下の2つだけ覚えてください。
- train()関数
- predict()関数
train関数でモデルを推定して、predict関数で予測を行います。
4.train関数の使い方
train()関数の引数として、とりあえず以下のものを覚えておくとよろしいかと思います。
- formula:モデルの書式
- data:トレーニングデータ
- method:使う手法
- tuneLengthまたはtuneGrid:パラメタチューニングの範囲
- preProcess:データの前処理
- trControl:その他のコントロール
順に説明します。
formula:モデルの書式
応答変数と説明変数の対応を指定します。
これは一般化線形モデル(glm関数)とまったく同じ書式で書くことができます。
機械学習は、パッケージによって、formulaの書き方が変わることも多いのですが、caretパッケージはそこを吸収してくれます。
例えば、応答変数がyで、説明変数にx1,x2,x3があれば、以下のように書きます。
y ~ x1 + x2 + x3
また、交互作用がある場合は、コロン(:)を使います。
交互作用については、統計勉強会の資料を参照してください。一番下に交互作用を説明したスライドがあります。
y ~ x1 + x2 + x3 + x1:x2
すべての変数を使う場合は、ピリオド(.)を使います
y ~ .
すべての変数で、2次の交互作用を使う場合は、2乗記号(^2)を使います。
y ~ (.)^2,
data:トレーニングデータ
トレーニング用のデータを指定します。
例えば、以下のように指定します。
data = dataTrain
method:使う手法
methodと呼ばれる引数を指定することによって、使う手法を変えることができます。
ランダムフォレストだろうが、ニューラルネットワークだろうが、この引数を変えるだけでOK。便利ですね。
ニューラルネットワークを「nnetパッケージ」で計算する場合には、以下のように指定します。
method = “nnet”
同じランダムフォレストでも、methodに”rf”、”ranger”、”Rborist”を指定することで、3種類のパッケージを使うことができます。
methodにはかなり多くの種類があります。
どのような手法を設定できるかは、以下のリンク先を参照してください(英語です)。
caretパッケージで指定できるmethod一覧
新しい手法を使うとき、以下のように聞かれることがあります。
1 package is needed for this model and is not installed. (パッケージ名). Would you like to try to install it now?
1: yes
2: no
これがコンソールに出てきたら、半角の「1」を入れてエンターキーを押してください。必要なパッケージが勝手にインストールされます。
tuneLengthまたはtuneGrid:パラメタチューニングの範囲
ハイパーパラメタのチューニングも、caretにお任せ。
私たちは、チューニングしたいパラメタの範囲を指定するだけでOKです。
チューニングする範囲が明確に決まっていない場合は「何通りのパラメタを適用するか」を指定します。
それがtuneLength 。以下のように、数値を指定します。
tuneLength = 4
method = “nnet”を指定した場合は、チューニングするパラメタが2種類あります。
各々4通りのパラメタを適用するため、4 × 4 = 16通りのパラメタの組み合わせが試行されます。
あまり細かくすると、計算に時間がかかるので注意。
範囲をもっと細かく指定したい場合は、以下のようにします(method = “nnet”を指定した場合)。
tuneGrid = expand.grid(size=c(1:10), decay=seq(0.1, 1, 0.1))
method = “nnet”を指定した場合はsizeとdecayという2つのパラメタがチューニングの対象となります。
それを各々、以下のように変えます。
size :1~10まで1ずつ変更。
decay :0.1~1まで0.1ずつ変更
すると10 × 10 = 100通りのパラメタの組み合わせとなります。
チューニングの対象となるパラメタが何なのかは、先ほども紹介したリンク先の資料に載っています。
caretパッケージで指定できるmethod一覧
preProcess:データの前処理
データの前処理をしたい場合も、引数に指定をするだけでOKです。
たとえば、データを正規化したいと思った場合は、以下のように指定します。
preProcess = c(‘center’, ‘scale’)
ほかにも様々な前処理ができます。
データの変換は、解釈の困難性を生むため、なるべく避けているのですが、正規化くらいはしておいても損はないと思います。
むしろやらなかったら、精度が落ちることも。
正規化に関しては、簡単にできるので、指定しておきましょう。
それ以外の前処理に関しては、データと相談して決めてください。
trControl:その他のコントロール
その他、いろいろ指定ができます。あまりにも高機能なためここでは紹介しきれませんが、とりあえず、予測の評価方法の指定の仕方を書いておきます。
クロスバリデーションを使う場合は、以下のように指定します。
trControl = trainControl(method = “cv”)
ほかにも、ブートストラップ法を使うならば、”cv”のところを”boot”に変更します。
なお、”cv”を指定したときは、デフォルトで、10 fold CVを実行しています。
これはデータを10等分に分けて、評価をする方法です。
例えば、100個のデータがあった時、最初のNo:1~10のデータをテスト用データにし、残りのNo11~100のデータトレーニングデータにします。
次は、No11~20をテスト用データにして、それ以外をトレーニングデータにする・・・を10回繰り返し。
そして、トレーニングデータで学習して、テスト用データで予測の評価を行います。
データの分割の仕方を変えたければ、trainControl(method = “cv”, number = 20)のように、numberを変更します。
あたりまえですが、分割数を増やすと、その分たくさん「学習~予測の評価」を繰り返すことになるので、時間がかかります。number=20なら、20回繰り返しになるわけですね。
分割数は、CPUの資源と相談して決めてください。
ここまでくれば、caretパッケージを使うことで、「予測手法の選択」「実装方法(使用パッケージ)の選択」「パラメタのチューニング」「予測の評価」が簡単にできることがご理解いただけるかと思います。
並列化演算による高速化
caretパッケージは大変高機能なのですが、計算量が膨大になり、時間がかかることもしばしば。
そんな場合は、計算を並列化して、計算速度を上げます。
# 並列化演算を行う
# install.packages("doParallel")
library(doParallel)
cl <- makePSOCKcluster(4)
registerDoParallel(cl)
並列化にはdoParallelパッケージを使います。
管理人の使っているSurface Pro4 はCore i 5なので、コア数は4つでした。
コア数がわからなければ、『detectCores()』というRの関数を実行すれば簡単にわかるようです(ksato様からの情報です。ありがとうございます)。
管理人のPCの場合、これをやるかやらないかで、計算時間が倍近く変わりました。
※コマンドプロンプトを起動して、以下のコマンドを打つことによっても、コア数を調べることができます。
「set NUMBER_OF_PROCESSORS」
なお、コマンドプロンプトは、Windowsボタン + r で「ファイル名を指定して実行」を呼び出し、 cmd と打ってからエンターキーを押せば出てきます(真っ黒な画面です)。ご自身のPCのスペックに合わせてmakePSOCKcluster(4)の関数の中の数値を変えてください。
5.予測の方法
予測は簡単。以下のように指定するだけです。
predict(作られたモデル, 予測対象データ)
スポンサードリンク
6.caretを使った回帰の例
お待たせいたしました。caretやってみます。
予測には、定量的に数値を予測するものと、グループを分けるものがあります。
前者が回帰問題、後者が分類問題と呼ばれることもあるので、caretの実践例も、回帰と分類に分けてみました。
なお、methodによっては、回帰しかできないor分類しかできないものもあります。
詳しくはマニュアルを参照して下さい。
Type: Classification, Regressionと書いてあれば両方OK.
Type: Classificationだけなら、分類のみ。
Type: Regressionだけなら、回帰のみしか使えません。
まずは、回帰をしてみます。
データのシミュレーション
高機能な機械学習法を適用するにふさわしい、非線形+交互作用ありのシミュレーションデータを作ります。
#---------------------------------------------------------------------------------- # シミュレーションでデータを作る:回帰 #---------------------------------------------------------------------------------- set.seed(0) N <- 1500 x1 <- runif(N, min = -5, max = 5) x2 <- runif(N, min = -10, max = 10) x3 <- runif(N, min = 0, max = 10) y <- (sin(x1 * pi/2)*4 + x1*0.5) * x2 + x3*2 + (x3^2)*0.4 + rnorm(N, mean = 0, sd = 1) # 図示 par(mfrow=c(3,1)) plot(y ~ x1, main="x1") plot(y ~ x2, main="x2") plot(y ~ x3, main="x3") par(mfrow=c(1,1))
sin()関数を使って、説明変数x1をグネグネと曲げさせた上に、x1とx2を掛け合わせて交互作用を入れてみました。
おまけとして突っ込んだx3も2乗項を入れてあります。
こんなグラフになります。これだけ見ると、x1が予測の役に立つようには見えませんね(グラフをクリックすると大きくなります)。
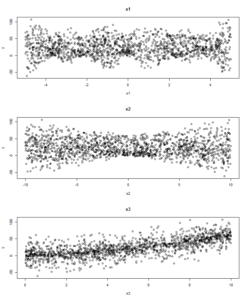
ちなみに、ノイズはとても小さくしてある(rnorm(N, mean = 0, sd = 1)なので、標準偏差1の正規乱数だけです)ので、実はかなり精度よく予測ができるはずのデータです。
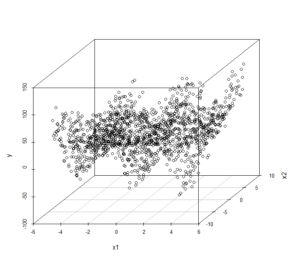
x1とx2の交互作用を見やすくするために、3次元プロットを描いてみます。
必要に応じて「scatterplot3d」パッケージをインストールする必要があるので注意してください。Rを右クリックして管理者として実行しないとパッケージがインストールできないことがあります。
# 3次元プロット
install.packages("scatterplot3d")
library(scatterplot3d)
scatterplot3d(x1, x2, y)
最後に、データをdata.frame形式にまとめて、さらに、トレーニング用データとテスト用データに分けます。
# data.frameにまとめる。 dataRegression <- data.frame( y = y, x1 = x1, x2 = x2, x3 = x3 ) # テスト用と学習用にデータを分ける dataRegressionTrain <- dataRegression[1:1000,] dataRegressionTest <- dataRegression[1001:1500,]
今回使うモデル一覧
今回はニューラルネットワークを1種類、ランダムフォレストをパッケージを変えて3種類試してみます。
さらに、比較のため、線形の重回帰分析も適用し、機械学習法がどれほど優れているかをご覧に入れます。
重回帰
まずは、重回帰分析をしてみます。
formulaに「y ~ (.)^2」と指定しているので、2次の交互作用まですべて入っています。
#---------------------------------------------------------------------------------- # 重回帰分析による予測 #---------------------------------------------------------------------------------- modelLm <- lm( y ~ (.)^2, data = dataRegressionTrain ) modelLm <- step(modelLm)
モデルを作った後で、step関数を使って変数選択をしてみました。
変数選択についてはこちらを参照してください。
最終的に選ばれた変数はこちらです。
意外と(?)ちゃんと変数選択できていました。
> modelLm Call: lm(formula = y ~ x1 + x2 + x3 + x1:x2, data = dataRegressionTrain) Coefficients: (Intercept) x1 x2 x3 x1:x2 -7.40147 0.04658 -0.09314 6.07932 0.51417
caretパッケージを使う下準備
次はいよいよ、caretパッケージを使います。
まずは、下準備。必要に応じてパッケージをインストールしてください。
#----------------------------------------------------------------------------------
# caretパッケージによる予測
#----------------------------------------------------------------------------------
# 必要であれば、caretパッケージをインストール
install.packages("caret")
library(caret)
# 並列化演算を行う
install.packages("doParallel")
library(doParallel)
cl <- makePSOCKcluster(4)
registerDoParallel(cl)
caretパッケージの使用と並列化の実行はほぼセットでよいと思います。
ニューラルネットワーク
ニューラルネットワークを実装してみます。
以下のコードは、管理人のPCで、実行に1~2分ほどかかりました。
# ニューラルネットワークによる予測
set.seed(0)
modelNnet <- train(
y ~ (.)^2,
data = dataRegressionTrain,
method = "nnet",
preProcess = c('center', 'scale'),
trControl = trainControl(method = "cv"),
tuneGrid = expand.grid(size=c(1:10), decay=seq(0.1, 1, 0.1)),
linout = TRUE
)
ほぼ説明済みかと思いますが、唯一、10行目の「linout = TRUE」には気を付けてください。
ここをFALSEにすると、分類をしようとしてしまいます。回帰をしてほしいので、TRUEを指定します。
この引数はnnetにのみ必要です。
結果はこちら。
一番良いパラメタを選んでくれました。
size = 10、decay = 0.9が一番いいらしいです。
> modelNnet Neural Network 1000 samples 3 predictor Pre-processing: centered (6), scaled (6) Resampling: Cross-Validated (10 fold) Summary of sample sizes: 900, 900, 900, 900, 900, 900, ... Resampling results across tuning parameters: size decay RMSE Rsquared 1 0.1 16.87378 0.5914678 1 0.2 16.88844 0.5907242 1 0.3 16.90042 0.5901307 1 0.4 16.91199 0.5895561 1 0.5 16.92221 0.5890620 1 0.6 16.93181 0.5885933 1 0.7 16.94099 0.5881513 1 0.8 16.94983 0.5877282 1 0.9 16.95842 0.5873208 1 1.0 16.96657 0.5869360 2 0.1 16.60414 0.6042189 ・・・中略 9 1.0 14.69518 0.6884453 10 0.1 15.13817 0.6731103 10 0.2 15.21062 0.6671152 10 0.3 14.98289 0.6793787 10 0.4 14.49030 0.6984681 10 0.5 14.24417 0.7107284 10 0.6 15.12805 0.6729213 10 0.7 14.64491 0.6936095 10 0.8 14.41727 0.7036555 10 0.9 13.96514 0.7187031 10 1.0 14.89217 0.6819798 RMSE was used to select the optimal model using the smallest value. The final values used for the model were size = 10 and decay = 0.9.
ランダムフォレスト
続いてランダムフォレストを実装します。
といっても、methodを変えるだけですが。
# ランダムフォレストによる予測
# randomForestパッケージを使う。
set.seed(0)
modelRF <- train(
y ~ (.)^2,
data = dataRegressionTrain,
method = "rf",
tuneLength = 4,
preProcess = c('center', 'scale'),
trControl = trainControl(method = "cv")
)
# rangerパッケージを使う。
set.seed(0)
modelRanger <- train(
y ~ (.)^2,
data = dataRegressionTrain,
method = "ranger",
tuneLength = 4,
preProcess = c('center', 'scale'),
trControl = trainControl(method = "cv")
)
# Rboristパッケージを使う。
set.seed(0)
modelRborist <- train(
y ~ (.)^2,
data = dataRegressionTrain,
method = "Rborist",
tuneLength = 4,
preProcess = c('center', 'scale'),
trControl = trainControl(method = "cv")
)
ここまでで、各々のモデルにおいて、パラメタのチューニングができたことになります。
パラメタのチューニングでは、「10 fold CVを使って評価された予測精度」が最もよくなるものが選ばれています。
次は、最適なパラメタを使って、予測モデルの手法(パッケージ)ごとの予測精度の比較をしてみましょう。
予測の評価
すべてのモデルを使って、テストデータを予測してみます。
上から順番に
重回帰
ニューラルネットワーク
ランダムフォレスト(RandomForestパッケージ)
ランダムフォレスト(rangerパッケージ)
ランダムフォレスト(Robristパッケージ)
です。
# テストデータで予測する predLm <- predict(modelLm, dataRegressionTest) predNnet <- predict(modelNnet, dataRegressionTest) predRF <- predict(modelRF, dataRegressionTest) predRanger <- predict(modelRanger, dataRegressionTest) predRborist <- predict(modelRborist, dataRegressionTest)
RMSEを計算してみます。これは誤差の大きさですので、小さければ小さいほどよく当たっていることになります。
> # RMSE > sqrt( sum((dataRegressionTest$y - predLm)^2) / 500 ) [1] 16.08433 > sqrt( sum((dataRegressionTest$y - predNnet)^2) / 500 ) [1] 13.06939 > sqrt( sum((dataRegressionTest$y - predRF)^2) / 500 ) [1] 7.035246 > sqrt( sum((dataRegressionTest$y - predRanger)^2) / 500 ) [1] 7.429744 > sqrt( sum((dataRegressionTest$y - predRborist)^2) / 500 ) [1] 7.694746
一番良いのは、ランダムフォレスト(RandomForestパッケージ)の7.035でした。
全体的に見ても、ランダムフォレストの予測精度が良いみたいですね。
というわけで、様々なモデル、パッケージ、パラメタを変えてみましたが、比較的短いコードで全部を実装して比較することができました。
caret便利だなと実感していただければ幸いです。
7.caretを使った分類の例
続いて分類です。
使うデータ
データは、よく使われるアヤメのデータを使います。
アヤメの種類を、花弁の大きさなどの特徴を使って、自動で分類します。
アヤメのデータはこちら。
> # アヤメのデータを使う。 > # アヤメの種類を判別する > head(iris) Sepal.Length Sepal.Width Petal.Length Petal.Width Species 1 5.1 3.5 1.4 0.2 setosa 2 4.9 3.0 1.4 0.2 setosa 3 4.7 3.2 1.3 0.2 setosa 4 4.6 3.1 1.5 0.2 setosa 5 5.0 3.6 1.4 0.2 setosa 6 5.4 3.9 1.7 0.4 setosa
データの番号が3の倍数のものをテストデータ。それ以外をトレーニング用データに分けます。
> indexIris <- which(1:nrow(iris)%%3 == 0) > indexIris [1] 3 6 9 12 15 18 21 24 27 30 33 36 39 42 45 48 51 54 57 60 63 66 69 72 75 78 81 84 87 90 [31] 93 96 99 102 105 108 111 114 117 120 123 126 129 132 135 138 141 144 147 150 > irisTrain <- iris[-indexIris,] > irisTest <- iris[indexIris,]
caretによるモデル化
続いて、モデル化とハイパーパラメタのチューニングをします。
ニューラルネットワークとランダムフォレスト(RandomForestパッケージ)のみを使います。
ニューラルネットワークの場合は、linout = Fとすることに注意してください。分類の場合はFALSEを指定します。
# ニューラルネットワーク
set.seed(0)
irisNnet <- train(
Species ~ (.)^2,
data = irisTrain,
method = "nnet",
tuneLength = 4,
preProcess = c('center', 'scale'),
trControl = trainControl(method = "cv"),
linout = F
)
# ランダムフォレスト(RandomForestパッケージ)
set.seed(0)
irisRF <- train(
Species ~ (.)^2,
data = irisTrain,
method = "rf",
tuneLength = 4,
preProcess = c('center', 'scale'),
trControl = trainControl(method = "cv")
)
これで、パラメタのチューニングまで終了です。
次は最適なパラメタを使って、テストデータを予測し、手法の比較を行います。
予測の評価
まずは、テスト用データを予測します。
# 予測 # ニューラルネットワーク predIrisNnet <- predict(irisNnet, irisTest) # ランダムフォレスト(RandomForestパッケージ) predIrisRF <- predict(irisRF, irisTest)
続いて、精度評価です。
分類問題の場合は、confusionMatrix()という関数を使うと簡単です。
> # 評価
> # ニューラルネットワーク
> confusionMatrix(data = predIrisNnet, irisTest$Species)
$positive
NULL
$table
Reference
Prediction setosa versicolor virginica
setosa 16 0 0
versicolor 0 14 0
virginica 0 3 17
$overall
Accuracy Kappa AccuracyLower AccuracyUpper AccuracyNull AccuracyPValue McnemarPValue
9.400000e-01 9.099640e-01 8.345181e-01 9.874514e-01 3.400000e-01 5.551972e-19 NaN
$byClass
Sensitivity Specificity Pos Pred Value Neg Pred Value Precision Recall F1 Prevalence Detection Rate Detection Prevalence Balanced Accuracy
Class: setosa 1.0000000 1.0000000 1.00 1.0000000 1.00 1.0000000 1.0000000 0.32 0.32 0.32 1.0000000
Class: versicolor 0.8235294 1.0000000 1.00 0.9166667 1.00 0.8235294 0.9032258 0.34 0.28 0.28 0.9117647
Class: virginica 1.0000000 0.9090909 0.85 1.0000000 0.85 1.0000000 0.9189189 0.34 0.34 0.40 0.9545455
$mode
[1] "sens_spec"
$dots
list()
attr(,"class")
[1] "confusionMatrix"
> # ランダムフォレスト(RandomForestパッケージ)
> confusionMatrix(data = predIrisRF, irisTest$Species)
$positive
NULL
$table
Reference
Prediction setosa versicolor virginica
setosa 16 0 0
versicolor 0 14 1
virginica 0 3 16
$overall
Accuracy Kappa AccuracyLower AccuracyUpper AccuracyNull AccuracyPValue McnemarPValue
9.200000e-01 8.799520e-01 8.076572e-01 9.777720e-01 3.400000e-01 1.281546e-17 NaN
$byClass
Sensitivity Specificity Pos Pred Value Neg Pred Value Precision Recall F1 Prevalence Detection Rate Detection Prevalence Balanced Accuracy
Class: setosa 1.0000000 1.0000000 1.0000000 1.0000000 1.0000000 1.0000000 1.0000000 0.32 0.32 0.32 1.0000000
Class: versicolor 0.8235294 0.9696970 0.9333333 0.9142857 0.9333333 0.8235294 0.8750000 0.34 0.28 0.30 0.8966132
Class: virginica 0.9411765 0.9090909 0.8421053 0.9677419 0.8421053 0.9411765 0.8888889 0.34 0.32 0.38 0.9251337
$mode
[1] "sens_spec"
$dots
list()
attr(,"class")
[1] "confusionMatrix"
たくさん情報が出てきてちょっと見づらいんですが、$overallのAccuracyを見ると、ニューラルネットが的中率94%。ランダムフォレストが92%でした。
今度はニューラルネットワークの方が精度が高くなっているようです。
8.caretを使って勾配ブースティング(Xgboost)
最後に、予測精度がとても高いことで有名な勾配ブースティングと呼ばれる少し発展的な手法を使ってみます。
発展的な手法であろうが、難しい手法であろうが、methodを変えるだけ。
やってみます。
Xgboostは、回帰でも分類でもどちらでも使うことができます。
今回は、回帰の時に使ったシミュレーションデータをそのまま使います。
xgBoostにはmethodが「xgbLinear」と「xgbTree」があります。
これらは、ともに同じパッケージを使っているのですが、計算の仕方が異なるので、2パタンで計算してみました。
#----------------------------------------------------------------------------------
# もっと複雑な手法を試してみる
#----------------------------------------------------------------------------------
# 勾配ブースティングによる予測 xgboost:線形予測の結合
set.seed(0)
modelXgboostLinear <- train(
y ~ (.)^2,
data = dataRegressionTrain,
method = "xgbLinear",
preProcess = c('center', 'scale'),
trControl = trainControl(method = "cv"),
tuneLength = 4
)
# 勾配ブースティングによる予測 xgboost:Treeモデルの結合
set.seed(0)
modelXgboostTree <- train(
y ~ (.)^2,
data = dataRegressionTrain,
method = "xgbTree",
preProcess = c('center', 'scale'),
trControl = trainControl(method = "cv"),
tuneLength = 4
)
結果はこちら。
かなりたくさんのパタンのハイパーパラメタの組み合わせを試してくれていることがわかります。
> modelXgboostLinear eXtreme Gradient Boosting 1000 samples 3 predictor Pre-processing: centered (6), scaled (6) Resampling: Cross-Validated (10 fold) Summary of sample sizes: 900, 900, 900, 900, 900, 900, ... Resampling results across tuning parameters: lambda alpha nrounds RMSE Rsquared 0.000000000 0.000000000 50 7.275041 0.9255692 0.000000000 0.000000000 100 7.210353 0.9269785 0.000000000 0.000000000 150 7.202916 0.9271017 0.000000000 0.000000000 200 7.201509 0.9271269 0.000000000 0.000100000 50 7.288270 0.9252462 ・・・中略 0.100000000 0.003162278 200 7.035043 0.9293826 0.100000000 0.100000000 50 7.091104 0.9282449 0.100000000 0.100000000 100 7.032328 0.9294651 0.100000000 0.100000000 150 7.025005 0.9295980 0.100000000 0.100000000 200 7.023799 0.9296224 Tuning parameter 'eta' was held constant at a value of 0.3 RMSE was used to select the optimal model using the smallest value. The final values used for the model were nrounds = 200, lambda = 0.1, alpha = 0.1 and eta = 0.3. > modelXgboostTree eXtreme Gradient Boosting 1000 samples 3 predictor Pre-processing: centered (6), scaled (6) Resampling: Cross-Validated (10 fold) Summary of sample sizes: 900, 900, 900, 900, 900, 900, ... Resampling results across tuning parameters: eta max_depth colsample_bytree subsample nrounds RMSE Rsquared 0.3 1 0.6 0.5000000 50 14.766303 0.6898520 0.3 1 0.6 0.5000000 100 14.525127 0.6988411 0.3 1 0.6 0.5000000 150 14.499788 0.7004231 0.3 1 0.6 0.5000000 200 14.485881 0.7009343 ・・・中略 0.4 4 0.8 0.8333333 100 6.704750 0.9357635 0.4 4 0.8 0.8333333 150 6.619909 0.9373976 0.4 4 0.8 0.8333333 200 6.599515 0.9377918 0.4 4 0.8 1.0000000 50 7.267796 0.9260153 0.4 4 0.8 1.0000000 100 6.984339 0.9313674 0.4 4 0.8 1.0000000 150 6.955028 0.9317949 0.4 4 0.8 1.0000000 200 6.936622 0.9321513 Tuning parameter 'gamma' was held constant at a value of 0 Tuning parameter 'min_child_weight' was held constant at a value of 1 RMSE was used to select the optimal model using the smallest value. The final values used for the model were nrounds = 200, max_depth = 3, eta = 0.3, gamma = 0, colsample_bytree = 0.8, min_child_weight = 1 and subsample = 1.
テストデータを予測します。
# 予測 predxgBoostLinear <- predict(modelXgboostLinear, dataRegressionTest) predxgBoostTree <- predict(modelXgboostTree, dataRegressionTest)
予測精度を評価します。
> # RMSEを、最高精度だったランダムフォレストと比較 > sqrt( sum((dataRegressionTest$y - predRF)^2) / 500 ) [1] 7.035246 > sqrt( sum((dataRegressionTest$y - predxgBoostLinear)^2) / 500 ) [1] 5.844164 > sqrt( sum((dataRegressionTest$y - predxgBoostTree)^2) / 500 ) [1] 4.610859
methodに”xgbTree”を指定したときに最高精度「4.611」となっています。
さすがというか、素晴らしい性能です。
ランダムフォレストの最高精度「7.035」を軽々下回る値が出ました。
今回は、変数選択を一切しませんでした。
交互作用はx1とx2の間にのみあり、ほかの変数の間にはありません。
そういった部分を修正すれば、さらに精度が上がる可能性もあります。
このように、たくさんの手法で何回も何回もモデルを試行錯誤して作っていく場合に、caretパッケージは大変便利だというお話でした。
参考文献
|
はじめてのパターン認識 名前の通り、初めて学ぶ人にとってちょうど良い入門書です。 機械学習とパターン認識の概要と、k最近傍法やニューラルネットワーク、サポートベクトルマシンにランダムフォレストといった各種法の概要など、幅広く載っています。 まずはこの本から機械学習を学ばれるという方は多いかと思います。 |
 |
|
データサイエンティスト養成読本 機械学習入門編 雑誌のような体裁の本です。機械学習の理論について細かく載っているわけではありませんが、大枠をつかむ入門資料として、良い本なのではないかと思います。caretについても、ほんの少しだけ載っています。 |
 |
樹木構造接近法 ランダムフォレストをはじめとしたTreeモデル関連について記載がある本です。私の修論はランダムフォレストだったのですが、この本には大変お世話になりました。Rのコードも多く、参考になります。 |
 |
|
ゼロから作るDeep Learning ―Pythonで学ぶディープラーニングの理論と実装 ニューラルネットについて勉強したければこの本がおすすめです。 2016年に読んだ本の中で、最も良い本でした。ぜひ一読をお勧めします。 |
 |
書籍以外の参考文献
スポンサードリンク

4.の「並列化演算による高速化」のところでコア数を知るためには、
detectCores()関数でも出来るかと思います
ksato様
コメントありがとうございます。
管理人の馬場です。
確かにその通りですね。情報ありがとうございます。
あきらかにこのやり方のほうが簡単であるため、記事もそのように修正しておきました。
ありがとうございます。
careについて調べており、貴サイトを訪問させていただきました。
caretもバージョンアップされていますので、最新のcaretを取り込んだ記事を期待しています。
caretでLightGBMを利用できるかどうかに興味があり、調べていますが、見つけることはできません。
もし、ご存知でしたら、ご教示いただけましたら大変に助かります。
notitake様
コメントありがとうございます。
管理人の馬場です。
おそらく現在、LightGBMは、引数では指定できないはずです。
ただし、別のパッケージを使ってLightGBMを利用できるようにし、その結果をcaretに渡すことでクロスバリデーションなどを比較的容易に行うことはできるかもしれません(やったことは無いです)。